R벤치마크 - 데이터 불러오기
by DANBI
R벤치마크 - 데이터 불러오기
최근 공공데이터 개방, 다양한 데이터 분석 경진대회 (kaggle.com, bigcontest.or.kr 등) 그리고 데이터 사이언스에 대한 관심상승에 따라, 많은 양의 데이터를 쉽게 접할 수 있습니다. 저 역시 데이터 분석가로서 활동하면서, 특히 매순간마다 방대한 양의 데이터가 축적되는 게임회사에서 데이터 분석 업무를 진행하다 보니, 업무에서도 종종 어마어마한 양의 데이터를 다루게 됩니다. 특히 딥러닝과 같이 데이터가 많으면 많을수록 좋은 기법을 적용할 때는, 이렇게 많아도 되나 싶을 정도의 데이터와 씨름을 하게 되죠.
데이터의 종류 (문자열, 숫자, 범주형, 연속형 등)에 따라 접근 방식을 달리 하듯, 데이터의 크기에 따라 다른 방법들을 적용해야 합니다. 마치 컴퓨터용 사인펜은 OMR 카드의 빈칸을 채우는 용도로는 적합하지만, 큰 면적 (e.g. 벽, 책상, 천장 등)을 칠하는 데에는 적합하지 않고, 큰 면적을 칠하는 데 쓰이는 스프레이, 페인트 붓 등은 OMR 카드의 작은 정답란을 채우는 데에는 적합하지 않은 것처럼 말이지요.
사실 사인펜으로 벽을 칠하는 것이 불가능한 것은 아닙니다. 단지 매우 오래 걸리기 때문에, 그 누구도 그런 비효율적인 방법을 시도하지 않을 뿐이죠. 마찬가지로, 적은 양의 데이터를 다루는 방법을 대용량 데이터를 다룰 때에 적용하는 것이 불가능한 것은 아닙니다, 단지 매우 오래 걸리기 때문에 효율성이 많이 떨어질 뿐이죠. 이런 대용량의 데이터를 처리하기 위해 저 같은 R아보(R만 아는 바보)가 Stack Overflow 및 다른 포럼들을 기웃거리고 삽질을 하다 보면 대용량 데이터를 처리하기 위해서는 나름 몇몇 해결책을 터득하게 됩니다. 첫 번째 해결책은 바로 데이터 샘플링입니다.
데이터 샘플링
사실 5조5천억 행의 데이터가 주어진다고 해도, 모든 데이터를 굳이 다 써야 하는 경우는 극히 소수입니다. 무작위추출 및 다른 샘플링 기법들을 활용하면, 적은 양의 데이터를 통해 대용량의 데이터를 이용한 분석과 비슷한 결과 또는 성능을 도출할 수 있습니다. 사실 이런 방식으로 일부(sample)의 결과를 가지고 모집단(population)에 대하여 추론하는 것이 추론 통계학의 근간이기도 합니다. 특히 요즘처럼 너무 많은 양의 데이터를 쉽게 접할 수 있는 경우에는, 주어진 데이터 전체를 사용하는 것보다는, 샘플링을 진행하여 작고 빠르게 이것저것 시도해보는 방법이 적합합니다. 마치 agile하게 prototype을 만들며 개발을 진행하는 애자일 소프트웨어 개발론처럼 말이지요. 일례로 작년 (2017) IEEE Conference on Computational Intelligence and Games의 일부로 진행된 게임 데이터 마이닝 대회에서는 100Gb가 넘는 양의 게임 로그 데이터가 제공되었는데요, 해당 데이터 세트를 필자의 개인 노트북으로 해당 전부 읽는 데에 약 8시간 이상 걸려 포기한 적도 있습니다. (물론 R에서 파일을 읽어 들이는 가장 느린 방법으로 시도하기는 했습니다, 파일 읽어 들이기에 대한 벤치마킹은 아래 내용 참조!). 이런 경우에는 주어진 데이터 전체를 사용하기보다는, 무작위 추출하여 작은 데이터 세트를 만들고, 추출된 작은 데이터 세트를 탐사분석하고 가공한 뒤, 해당 프로세스를 전체에 적용하는 등의 방식을 취하는 것이 효율적일 것입니다. 데이터 샘플링 다음으로 대용량 데이터를 처리할 때 고려해야 하는 점은 바로 “대용량 데이터 전용 도구를 사용”하는 것입니다.
대용량 데이터 처리 도구
여기에서 도구는 대용량의 데이터를 처리하는 데 쓰이는 언어, framework등 모든 것을 총망라하는 단어입니다. Hadoop, 클러스터 이용, SQL기반 DB시스템 사용 등 무궁무진한 도구들이 존재하지만, 모든 사람에게 친숙하지는 않습니다. 특히 주로 개인 컴퓨터를 사용하여 데이터를 다뤄왔던 학생이나 개인에게, Hadoop을 설치한다거나 AWS의 클러스터를 설정하여 사용하는 것은 매우 힘든 일입니다. 저도 개인적인 목적을 위해 AWS의 free-tier cluster를 설정하여 작업을 진행해봤는데요, 공짜인 이유가 있었습니다. (매우 느렸습니다). 
대용량 처리 도구를 사용하는 것이 옳은 것을 안다고 해도, 설치하고 세팅하고 실제 사용하더라도 속도가 빠르다는 보장이 없기 때문에, 사용이 망설여집니다. 물론 저처럼 회사에서 남이 다 구축해놓고, 유지까지 해주면 편하게 사용할 수 있겠지만요. 대용량 데이터 처리 도구가 있는 것은 알지만, 저와 같은 R아보(R만 아는 바보)가 집에서 개인적인 프로젝트로 데이터를 다루면, 쌩 R로 작업을 하게 되는 경우가 대부분입니다. 인터넷의 포럼들을 기웃거리다 보면 앞서 말씀드린 샘플링 그리고 대용량 처리용 도구를 사용하는 것 외에도 많은 방법을 제시하고 있습니다. 더 좋은 컴퓨터를 사라는 물질만능주의의 팁도 있지만, R로 대용량 데이터를 다룰 때 이런저런 방법을 사용해보라고 얘기해주기도 하죠. 본 글은, “R로 대용량 데이터를 다룰 때 이런저런 방법”에 대해서 앞으로 얘기해보고자 합니다. 정확히는 필수불가결적인 요소로 R을 이용할 수 밖에 없는 상황일 때, 대용량의 데이터를 좀 더 빨리 효율적으로 가공하는 방법에 대하여 정리하고자 합니다. (절대, R외에 Python, SAS, MATLAB등의 다른 언어 및 프로그램들을 사용할 줄 몰라서 R만 쓰는 것이 맞습니다). 앞으로, R에서 사용하는 데이터 가공법 그리고 불러오기 방법들을 실험하여, R아보도 대용량 처리를 R에서 하는 방법에 대해서 R아보도록 하겠습니다. 먼저, 오늘은 데이터를 불러오는 방법에 대하여 글을 써보고자 합니다.
R에서 대용량 데이터 불러오기
데이터 가공에 앞서, 데이터를 불러오는 방식에 따른 성능 측정을 진행하여, 가적 효율적인 데이터 불러오기 방법을 찾아보고자 합니다. 앞서 언급했듯이, 최근에는 대용량의 데이터를 쉽게 접할 수 있지만, 종종 방대한 데이터를 어떻게 불러들이고 처리해야 할지 막막한 때도 있습니다. 특히 데이터를 먼저 엑셀로 살펴보는 방식을 취하시는 분들도 종종 있는데요, 이런 경우 파일의 크기가 1Gb이상인 경우 엑셀이 읽는 데에만 수십분이 걸리기도 합니다. 사용하는 방법에 따라 데이터를 읽어들이는데 걸리는 시간은 천차만별입니다. 따라서 위에서 만든 1000만 명의 신상정보 파일을 이용하여, 읽어들이는 파일의 크기와 읽는 방법에 따른 소요시간을 측정하여, 가장 효율적으로 데이터를 불러오는 방법을 찾아보도록 하겠습니다. 컴퓨터의 상태와 같은 알 수 없는 noise에 의해 소요시간이 영향을 받을 수 있으므로, 각 방법 및 데이터 크기별로 데이터 불러오기를 10번씩 진행, 소요시간의 중간값으로 벤치마킹을 진행하였습니다. 자세한 실험 방법은 아래와 같습니다. (참고로, R은 메모리의 용량에 많이 영향을 받는 언어입니다. 그렇기 때문에, 아래의 실험들은 메모리의 용량은 충분하다는 가정하에 진행하였습니다. 메모리가 부족한 경우, 가상 메모리로 대체하는 방법, 메모리를 더 사서 꼽는 방법 등이 존재하지만, 이에 대해서는 다른 글에서 다루도록 하겠습니다)
실험 방법
-
R에서 효율적인 데이터 가공 및 불러오기 방법을 알아보기위해, 다양한 크기와 방식으로 데이터를 다루고, 이때 걸리는 시간, 즉 런타임을 기준으로 효율성을 측정하였습니다. 런타임 측정 단위는 "초"입니다.
-
데이터 구성
-
이 벤치마크를 위해 사용될 가짜 데이터는 10000000 x 7 (1000만 행, 7열)는 아래의 방식으로 생성하였습니다.
-
birthyear=sample(1900:2018, size= 10000000,replace=TRUE) gender=sample(c("Male","Female"), size=10000000, replace=TRUE) names=sample(unique(babynames$name), size=10000000, replace=TRUE) #babynames 라이브러리 설치 필요 zipcode=as.numeric(sprintf("%05d", sample(0:9999,size=10000000, replace=TRUE))) married=sample(c(0,1), size=10000000, replace=TRUE) telephone=sprintf("%03d-%04d-%04d", sample(0:999, replace=TRUE, size=10000000), sample(0:9999, replace=TRUE, size=10000000), sample(0:9999, replace=TRUE, size=10000000)) ip.address=sprintf("%d.%d.%d.%d", sample(0:999, replace=TRUE, size=10000000), sample(0:999, replace=TRUE, size=10000000), sample(0:999, replace=TRUE, size=10000000), sample(0:999, replace=TRUE, size=10000000)) fake.data=data.frame(name=names, birthyear=birthyear, gender=gender, zipcode=zipcode, married=married, telephone=telephone, ip_address=ip.address) - 해당 데이터 저장시에는 csv의 형태로 저장하였습니다.
-
-
데이터 불러오기
- 위에서 생성된 데이터를 이용하여, 10, 100, 1000, 10000, 100000, 1000000, 10000000행의 파일을 생성
- 파일 불러오기 방식 별로 상기 생성된 파일들을 읽어온다
- 파일을 한번 읽는데 걸린 시간을 측정하며, 이를 10번 측정하여 소요시간의 중간값으로 효율성을 비교한다
read.table
R의 내장 기본 함수이면서 가장 많이 쓰이는 read.table의 속도를 측정하여, 이를 벤치마킹의 기준점으로 삼도록 하겠습니다. read.table은 sep, nrows, colClasses 등의 다양한 하이퍼 파라미터들을 사용하며, 이 하이퍼 파라미터들의 사용 여부에 따라 성능에도 큰 차이가 있습니다. 다양한 파라미터들이 존재하는 만큼, 아래의 파라미터들의 지정 여부에 따른 데이터 불러오기 성능을 측정 하였습니다.
성능 측정
1) 파라미터 미지정
기준점으로서의 역할을 수행할 수 있도록, “header=TRUE”외 다른 하이퍼 파라미터들은 기입하지 않은 채로 데이터를 불러오는 데 걸리는 시간을 측정하였습니다..
read.table(file, header=TRUE)
2) 열구분자 (sep)
read.table함수의 sep 파라미터는 값 구분자 (value separator)를 의미합니다. 한 줄(행)에 여러 값이 존재할 때, 이 값들을 구분할 수 있게 하는 특수값을 구분자라고 하지요. 각각에 해당하는 열로 인식하게 합니다. 값 구분자가 쉼표인 경우, 3열의 테이블은 다음과 같이 표현할 수 있습니다.
또한, 자주 접하게 되는 csv (comma separated values), tsv (tab separated values) 파일들은 각각 쉼표와 탭으로 값들을 구분된 텍스트 파일을 의미합니다. 구분자를 정확하게 명시하지 않으면, 다수의 열이 존재하는 테이블을 1열로 인식하여 읽게 되는 등의 문제가 생길 수 있습니다. 이런식으로 잘 못된 구분자가 사용된 경우의 데이터 불러오기 성능 또한 측정하였습니다.
read.table(file, header=TRUE,sep=",") #구분자 = 컴마 (,)
read.table(file, header=TRUE,sep="\t") #구분자 = 탭 (\t)
3) 열별 데이터 유형 (colClasses)
시험의 유형 (객관식인지 주관식인지, 시험시간, 문제 개수 등)을 알고 공부한 학생과, 아무것도 모르는 상태에서 공부한 학생의 시험 준비 효율성은 당연히 차이가 날 수밖에 없겠죠? 이는 데이터 불러들일 때에도 마찬가지입니다. 열마다 어떤 유형의 데이터 (숫자, 문자, Factor 등)이 주어질 예정인지, read.table 함수의 colClasses 파라미터를 통해 설정할 수 있으며, 이 파라미터의 설정 여부에 따른 성능을 측정하였습니다.
read.table(file, header=TRUE, sep="," ,
colClasses=c("character", #이름(names)
"numeric", #생일(birthyear)
"character", #성별(gender)
"numeric", #우편번호(zipcode)
"numeric", #결혼상태(married)
"character", #전화번호(telephone)
"character" #ip주소(ip_address)
))
4) 문자열 -> factor (stringsAsFactors)
문자열 데이터 각각이 의미를 지닐 때에는 (i.e. 성별, 국적, 거주지역 등), 문자열들을 범주형 변수로 간주해야 하며, 이때 “factor”로 지정을 하게 됩니다. 하지만 모든 문자열 데이터들이 범주의 성격을 지니고 있지 않습니다. 특히 본 글에서 사용하는 데이터의 경우, 이름, 전화번호, 아이피 주소는 추가 가공 없이는 사실상 아무런 정보를 내포하지 않는 단순한 문자열인데요, 이런 경우에는 범주형 데이터가 아닌 문자열 데이터로서 받아들이는 것이 적합합니다. 하지만 R에서의 data.frame을 다루는 대부분 함수 (데이터 불러오기 포함)들은 문자열 데이터를 범주형으로 인식하는 stringsAsFactors라는 파라미터를 기본적으로 사용하게 되어있습니다(stringsAsFactors=TRUE). 그러므로 열별 데이터 유형을 직접적으로 문자열로 지정한 경우 를 제외한 모든 방식들 또한 문자열 데이터를 factor로 불러오게 되며, 해당 파라미터 설정에 따른 성능의 차이를 확인해 보았습니다.
read.table(file, header=TRUE, sep=",", stringsAsFactors=FALSE)
5) 행 크기 (nrows)
열별 데이터 유형이 시험의 유형이라면, nrow는 시험 범위라고 할 수 있습니다. 컴퓨터에게 “앞으로 x줄의 데이터를 읽어들일꺼야”라고 알려주는 것이죠. 이에 따른 성능 역시 측정하였습니다.
read.table(file, header=TRUE, sep=",", nrows=x)
6) 모든 파라미터 지정
여지껏 언급된 모든 파라미터들을 설정하여, read.table을 이용한 최상의 데이터 불러들이기 속도를 확인해보았습니다.
성능 측정 결과
앞서 말씀드린 실험 방식으로 생산된 데이터들을 각각 10, 100, 1,000, 10,000, 100,000, 1,000,000, 10,000,000행의 파일로 저장하였고, read.table의 다양한 파라미터 설정 여부에 따른 각 파일들을 불러오는 속도를 측정 한 결과는 아래와 같습니다.
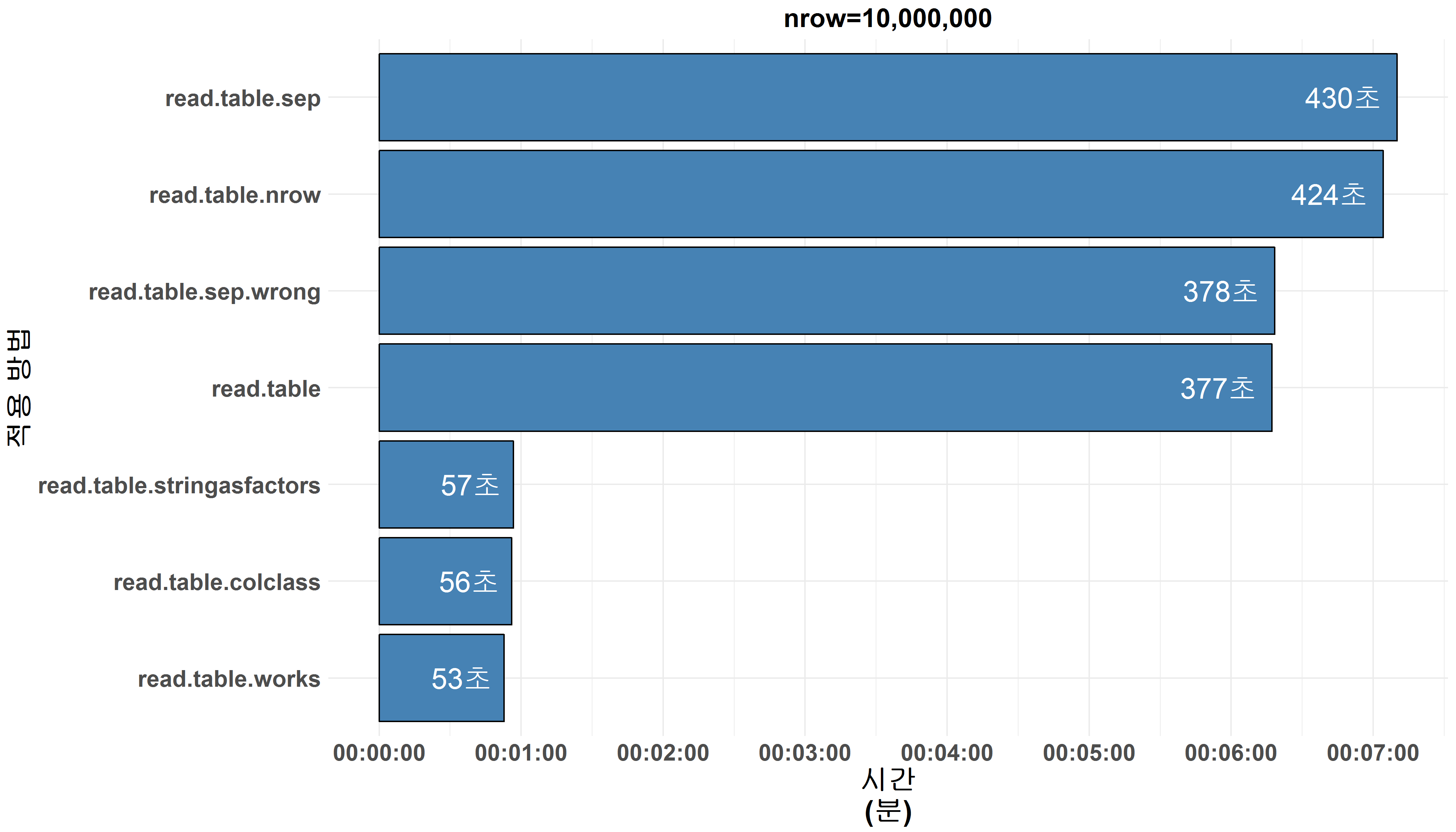
위 그래프는, 실험에서 사용된 가장 큰 크기의 천 만 행의 데이터를 불러올때 방법별 소요시간을 나타내며 보시는 바와 같이, 파라미터 설정 방식에 따라 큰 성능차이를 보여주고 있습니다. 1분이하의 소요시간을 보여준 파라미터 설정 방식 (빨간색 표기)이 존재하는 반면 6분 심지어 7분이상이 걸린 방법들이 존재합니다.
파라미터 미지정시, 천만행을 불러오는데 약 377초가 걸렸으며, 놀랍게도 이는 7개의 측정 대상방법 중 중간정도에 해당합니다. 하지만, 파라미터 미 지정시에는 열 구분자가 정상적으로 인식되지 않아, 해당 데이터가 갖고있는 형태 (10,000,000 행 x 7열)가 유지되지 않았습니다 (1열의 데이터로 인식됨). 그렇기 때문에 이 방법은 사실 정상적으로 데이터 불러오기가 수행되었다고 할 수 없겠습니다.
데이터의 형태 유지에 필수적인 올바른 열 구분자 지정하여 같은 데이터를 불러오는데 약 430초가 걸렸으며, 본 글에서 측정한 데이터 불러오기 방식 중 가장 느린 속도를 보여주었습니다.이는 각 행을 1열로 인식하고 불러들였던 위의 방식과는 달리, 1행당 7개의 값을 불러들였기 때문입니다. 오래 걸리는 대신, 정확하게 원 데이터의 형태 그대로 불러오기가 진행된 것이죠. 덕분에 데이터의 형태가 유지되어, 추가 작업 없이 데이터 열람 및 가공이 가능합니다. 잘못된 열 구분자를 부여한 경우, 파라미터 미지정한 경우와 거의 비슷한 속도 (377초 vs 378초)를 보여주었으며, 데이터의 형태가 유지되지 않아 1열의 데이터를 읽어들였습니다. 이를 통해 read.table 함수의 열구분자가 속도에 큰 영향을 미친다는 것을 확인 할 수 있습니다.
열별 데이터 유형 (colClasses)을 설정한 경우, 약 6배 정도 향상된 속도를 확인 할 수 있었으며 (56초 vs 430초), 이를 토대로, 열별 데이터 유형을 설정 해주는 것이 read.table 함수의 성능을 크게 향상시키는 것으로 보입니다. 하지만, 이 성능 향상의 원인은 사실 열별 데이터 유형 설정이 아닌 factor 미사용에 있습니다. colClasses를 지정하지 않는 경우, 숫자가 아닌 모든 열의 데이터는 문자열(character)로 간주하게 됩니다. 또한 R은 기본적으로 모든 문자열을 factor라는 데이터 유형으로 인식하도록 설정되어 있기 때문에, 모든 비숫자열의 데이터는 factor로 인식됩니다.
factor는 문자열 값과 그에 상응하는 범주를 나타내는 레벨값을 동시에 보유하는 데이터 유형입니다.
위의 코드와 같이, “서울”, “서울”,” 판교”, “제주도”로 구성된 벡터를 factor로 변경하면, 주어진 문자열값과 더불어 해당 벡터의 고유값에 해당하는 “서울”, “판교”, “제주도”라는 level이 생성됩니다. 또한 이 level을 토대로 1,1,3,2라는 벡터로 (i.e. level-1, level-1, level-3, level-2) 변경되어 저장되는 것을 확인 할 수 있습니다. 이렇게, factor는 주어진 벡터 (본문의 경우 한열에 저장된 모든 데이터 값)들의 고유값들에 따라 level을 생성, 재구성하여 저장하기 때문에 뒷선에서 문자열보다 많은 연상을 요구하는 데이터 유형입니다. 그렇기 때문에, 이 유형으로 데이터를 불러들이는 경우와 그렇지 않는 경우 간 큰 속도 차이를 보이게 됩니다 (56초 vs 430초). 각 열마다 데이터 유형을 설정하는 경우, 문자열들을 불러들일 때에 stringAsFactors=FALSE라고 지정한 것과 같으며, factor구성에 요구되는 추가 연산을 방지, 샹상된 속도를 보이게 되는 것입니다.
행 개수를 지정하여 데이터를 불러들이는 경우, 천 만행 불러오기 기준으로 미미한 속도 향상을 보여주었습니다 (424초 vs 430초). 마지막으로, 여지껏 언급된 read.table함수의 모든 파라미터들을 설정 한 결과, 천 만행의 데이터를 약 53초만에 읽어들이는 좋은 성능을 보여주었습니다.
데이터의 규모를 달리하여, 각 방법별로 성능을 측정해보니, 흥미로운 패턴을 확인 할 수 있었습니다.
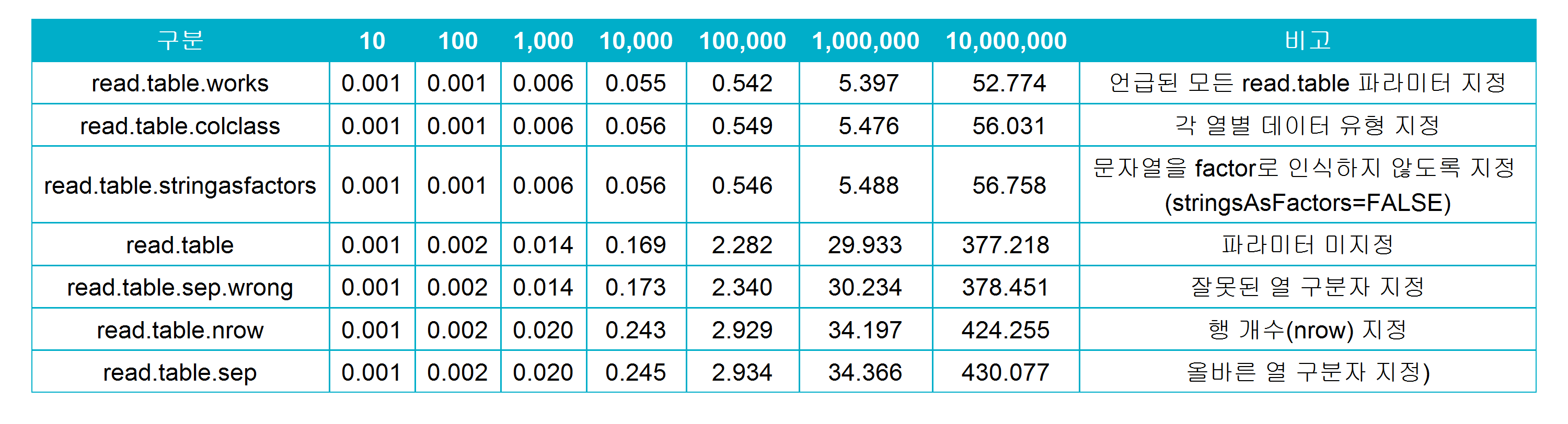
위의 표가 나타내는 것과 같이, 특정 파라미터 설정 여부와 상관없이, 행의 개수가 10배씩 증가함에 따라, 소요시간 역시 대략 10배 증가하는 것을 확인 할 수 있습니다. 심지어 가장 빠른 속도를 보였던 모든 파라미터 설정 방식 (read.table.works)조차, 행의 개수가 만, 십만, 백만 ,천만으로 증가함에 따라 각각 0.056초, 0.54초, 5.4초, 53초로 데이터 크기 증가율에 비례하는 소요시간을 보입니다. read.table 함수의 이런 특성 (데이터 크기 증가에 정비례하여 소요시간 또한 증가)을 이용하여, 데이터 불러오기 전 소요시간을 예상 할 수 있습니다.
read.table의 파라미터 설정에 따른 성능을 측정해본 결과, read.table사용시에는 그 어떤 파라미터보다 colClass를 지정하는 것, 그리고 factor 사용을 지양하는 것이 중요해 보입니다. 그렇다면, 다른 데이터 불러오기 함수들의 성능은 어떨까요?
read_csv 및 fread
read_csv(readr)
RStudio의 Import Dataset메뉴를 이용하여 데이터를 불러오는 경우, readr라이브러리의 read_csv함수를 사용하게 됩니다. readr라이브러리는 ggplot2, tidyr, dplyr 등 R세계에서 가장 유명한 라이브러리들을 만든 tidyverse팀이 작성한 라이브러리입니다. 쉽고, 실용적이며, 높은 효율성을 보이는 라이브러리들을 많이 만드는 팀인 만큼, read_csv역시 높은 성능과 실용성이 기대됩니다. 아래의 코드로 데이터 불러오기 성능을 측정하였습니다
read_csv(file)
fread(data.table)
data.table은 대용량 데이터를 다룰 때 극상의 성능을 보이는 것으로 널리 알려졌는데요, 데이터를 불러올 때도 마찬가지로 상당한 성능을 보여 줄 것으로 기대합니다. data.table라이브러리의 다양한 함수들이 최강의 성능을 보이는 이유는 메모리에 정보를 저장하는 방식이 다른 함수들과 다르기 때문이라고 합니다. read.table이나 다른 함수들은 buffer를 통해 메모리에 데이터를 저장하지만, fread는 포인터를 이용하여 메모리에 직접 데이터를 저장한다고 하니, 마치 중간 유통단계를 생략한 농수산물 직거래처럼 상당한 성능이 기대됩니다.
fread(file)
성능 측정 결과
앞서 진행한 성능 측정과 동일한 방식으로 read_csv와 fread 함수들의 데이터 읽기의 속도/성능을 측정한결과, 두 함수 모두 read.table에 비해 월등한 성능을 보여주었습니다. 약 1분 (53초)가 필요했던 read.table에 비해, 두 함 수 모두 30초 이내로 천 만 행의 데이터를 읽어들였습니다.
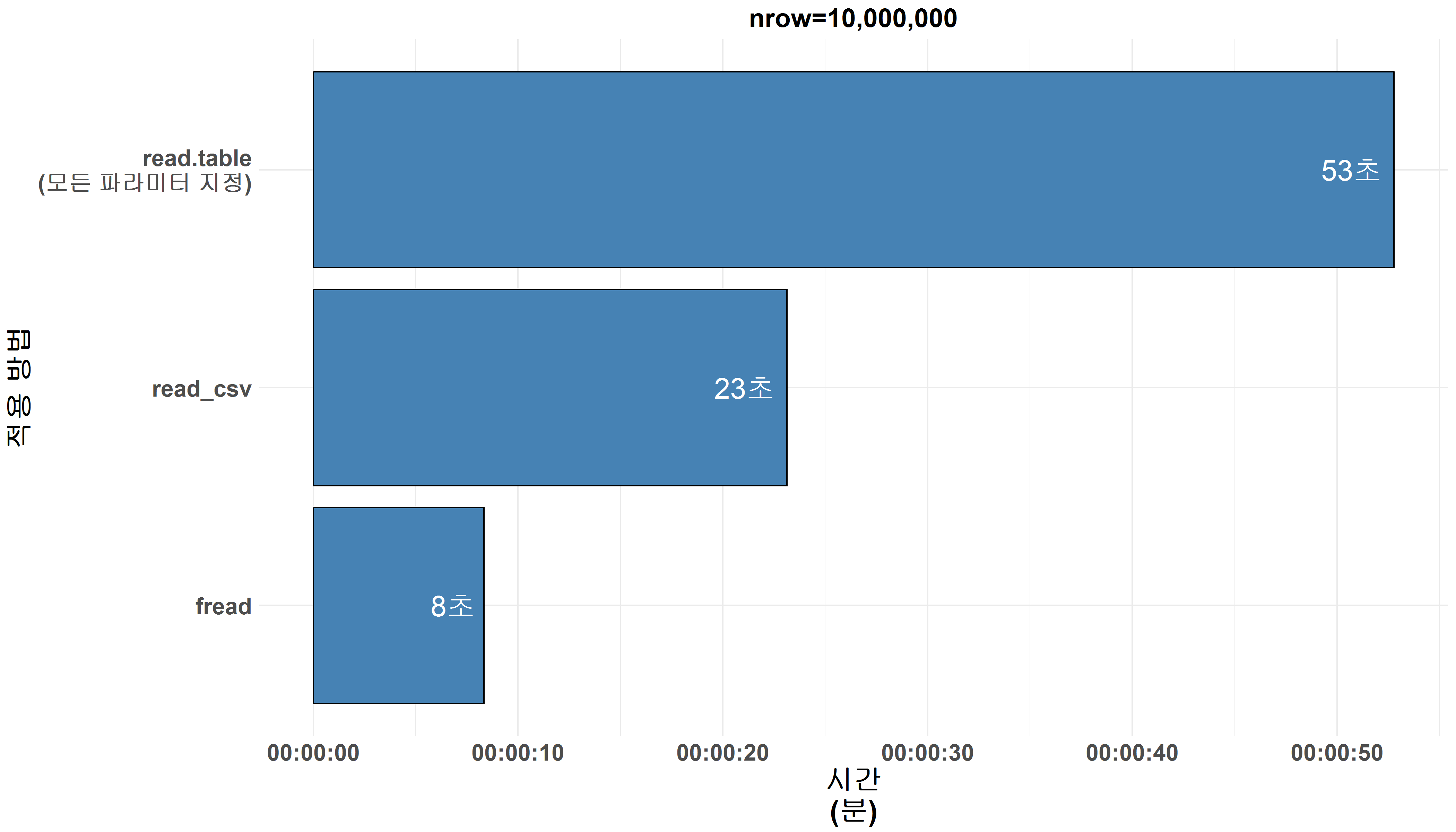
read_csv는 위에서 성능을 측정한 read.table 대비 약 2~3배 향상된 데이터 불러오기 속도를 보입니다 (23초 vs 53초). 추가로 read_csv는 read.table과는 달리, 열별 데이터의 유형을 지정해 주었을 때도 차이를 보이지 않았습니다. read.table은 관련 파라미터 미지정시 모든 열을 문자로 그리고 factor 인식하지만, read_csv는 데이터 유형 미지정 시 자동으로 첫 1,000줄을 기반으로 열별 데이터 유형을 지정하기 때문입니다.
fread는 read_csv 및 read.table보다 월등한 성능 (read_csv의 약 3배, read.table의 약 7배, 천만 행 데이터 기준 )을 보여주며 기대에 부응하였습니다. 그리고 fread 또한 read_csv 처럼 자동으로 열의 데이터 유형을 인식하여 올바른 데이터 유형으로 데이터를 저장하는 기능을 갖고있습니다. 한가지 유의해야 할 점은, fread 함수를 이용하여 데이터를 불러오는 경우, 결과물은 data.frame이 아닌 data.table의 형태를 띄게 됩니다. 두 유형간의 차이는 거의 없지만, 간혹 문제가 생기는 경우도 있기 때문에, data.frame으로 데이터 불러오기를 원하시는 경우에는 아래와 `같이 data.table=FALSE를 지정해주셔야 합니다.
fread(file, data.table=FALSE)
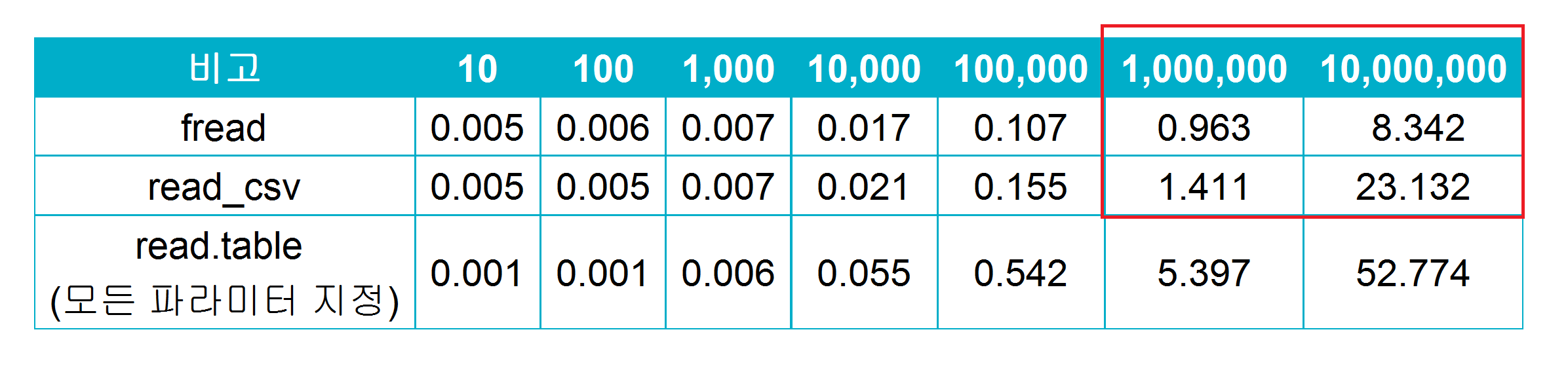
위의 표를 참조하면, read_csv함수의 경우 데이터의 크기 10배 증가에 따라, 소요시간은 약 16배 증가한 것을 알 수 있습니다. 반면, fread함수의 경우에는 오히려 소요시간은 약 9배만 증가했습니다 (백 만행 -> 천 만행 증가시, 빨간색 표기). 이를 토대로, read_csv함수는 read.table 함수보다 빠르지만, 데이터 크기 증가율을 상회하는 소요시간 증가율을 갖는 것을 알 수있으며, 이는 read_csv는 거대한 데이터 (본문에서 사용한 데이터보다 큰 규모의 데이터)를 불러들이는데에는 적합하지 않을 것으로 판단됩니다. 한편, fread는 데이터 크기 증가율 대비 소요시간이 적게 증가하였기 때문에, 대용량 데이터를 불러들이는데에도 적합 할 것으로 예상됩니다.
추가로, read_csv 및 fread 함수들의 경우 앞서 언급한 것과 같이 자동으로 각 열의 데이터 유형을 인식합니다. 그렇기 때문에, 한 열에 여러 유형의 데이터가 존재하거나, 오기입된 값이 있는 경우에는 오류가 발생할 수 있다는 점을 유의하셔야 합니다.
마무리
본 글에서 사용한 데이터 기준, 가장 빠른 속도를 보인 데이터 불러오기 방법은 data.table 라이브러리의 fread 함수였으며, 두 번째로 빠른 속도를 보인 read_csv 함수보다 약 3배 빠른 속도를 보여주었습니다. 또한, 가장 널리 쓰이는 R의 기본 데이터 불러오기 함수인 read.table은 파라미터 설정 여부에 따라 큰 성능 차이를 보였고, 특히 열별 데이터의 유형 지정 및 문자열 데이터를 factor로 불러오지 않는 것이 불러오기 속도에 큰 영향을 끼치는 것으로 확인되었습니다. R에서 대표적으로 데이터 불러오기에 쓰이는 함수와 함수들의 하이퍼 파라미터 설정에 따른 성능을 측정, 비교해보았습니다. 본 글에서 사용한 데이터의 경우에는, 10,000줄까지는 모든 함수들이 1초 이하의 런타임을 보였기 때문에, 큰 속도의 차이를 체감하기 어려울 것으로 보입니다. 하지만, 데이터의 크기가 증가함에 따라, 성능의 차이는 확연하게 느껴질 것으로 예상됩니다 (8초 vs 7분 10초). 그렇기 때문에, 작업 대상의 크기에 따라 적합한 함수를 선택하는 것이 업무 효율 증대에도 큰 도움이 되리라 생각합니다.
